微生物在哺乳动物肠道中定植跨物种遗传机制研究
摘要:尽管肠道微生物具有基础重要性,但其定植的遗传基础在很大程度上仍未得到探索。本文通过在生命之树尺度上应用跨物种基因型-生境关联分析,鉴定了与肠道定植相关的保守微生物基因模块。在数千个物种中,我们发现了79个分类学上多样化的推定定植因子,这些因子被组织成操纵子和非操纵子模块。它们包括先前已表征的定植途径,如自诱导剂-2生物合成,以及新过程,包括tRNA修饰和翻译。体内功能验证显示,YigZ(IMPACT家族)和tRNA羟基化蛋白-P(TrhP)是大肠杆菌肠道定植所必需的。仅过表达YigZ就足以将定植能力差的大肠杆菌MG1655的定植能力增强超过100倍。此外,YigZ的自然等位基因变异影响菌株间的定植效率。我们的发现突显了大规模比较基因组学在揭示微生物适应遗传基础方面的力量。这些广泛保守的定植因子可能对理解胃肠道失调和开发治疗方法至关重要。
背景本研究旨在系统揭示微生物在哺乳动物肠道中定植的遗传基础。尽管肠道微生物对人类健康至关重要,但其定植机制仍不明确,尤其是跨物种的共性遗传因素。先前研究多局限于少数可培养物种(如拟杆菌属),且难以覆盖不可培养或遗传难操作的微生物。随着宏基因组数据的积累,本研究开发了一种计算框架,通过在生命之树尺度上比较肠道与环境微生物的基因组,识别保守的定植相关基因模块,并辅以实验验证,以揭示广泛适用的定植机制。
文章内容总结
1.生命之树尺度的基因型-生境关联分析揭示保守的推定定植因子通过构建计算框架,从约28万个初始基因组中筛选出9,475个高质量、低冗余的基因组,并利用全对全蛋白质比对和互信息分析,系统性地寻找与哺乳动物肠道定植显著相关的蛋白质,成功鉴定出79个在系统发育上多样且与肠道环境显著相关的保守蛋白质家族,即推定定植因子,这些CFs在不同数据集和技术参数下均表现稳健。
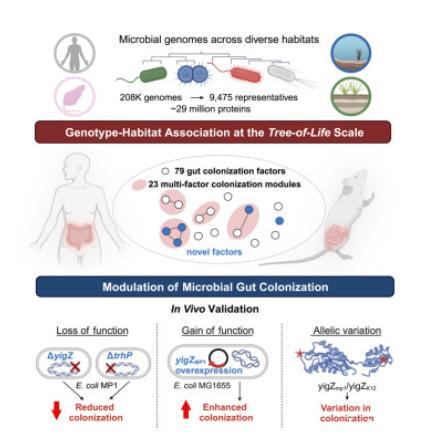
2.人与鼠肠道微生物的定植因子高度重叠通过比较在人类和鼠类肠道微生物中鉴定出的CFs,分析它们在不同宿主和基因组类型中的分布情况,发现37个CFs是人与鼠共有的,表明存在跨宿主的共同定植机制,同时部分CFs显示出对培养条件或宏基因组技术的偏好性。
3.定植模块揭示功能相关的基因群基于CFs在基因组中的共继承模式将它们分组为更高层次的功能单元——定植模块,最终确定了47个定植模块,其中23个包含多个CFs,这些模块既包括已知的操纵子结构,也揭示了新的非操纵子功能关联,表明CFs在基因组中是以功能协同的方式组织起来的。
4.定植模块涵盖已知和全新的生物学过程对CMs进行系统的功能注释以了解它们所代表的生物学过程,发现CMs的功能包括已知与定植相关的代谢、群体感应等过程,但也包含大量功能未知的蛋白质,提示存在此前未被认识的定植机制。
5.翻译相关因子是关键的定植模块通过深入分析得分最高的CMs,发现其中许多与tRNA修饰和翻译调控有关,从而将翻译相关过程确立为一类新的、与肠道定植密切相关的核心功能,这不同于传统的与厌氧代谢等相关的定植因子。
6.TrhP和YigZ被实验验证为大肠杆菌的必需定植因子在定植能力强的大肠杆菌MP1菌株中敲除四个选定的翻译相关CF基因,并通过与野生型菌株在小鼠肠道内竞争以评估其定植能力,结果显示敲除yigZ或trhP会显著削弱菌株的定植能力,而在体外不影响生长,证明它们是体内定植特异性的必需因子。
7.过表达YigZ足以显著增强弱定植菌株的定植能力通过在定植能力弱的大肠杆菌MG1655菌株中引入来自强定植菌株MP1的CF基因,测试其是否能"补救"MG1655的定植缺陷,发现仅过表达yigZ即可使MG1655的早期定植水平提升超过100倍,证明该单个基因在增强定植能力上具有充分性。
8.肠道微生物拥有更丰富的定植模块库通过比较肠道来源与环境来源的微生物基因组所携带的CMs数量,发现肠道微生物编码的CMs数量显著多于其环境中的近亲,表明成功的肠道定植者普遍依赖于一个更大的"定植工具包"。
9.定植因子在生命之树中广泛分布但模式各异将CFs的分析扩展至GTDB数据库的11.3万个代表性基因组,以考察其跨门类分布和系统发育信号,发现CFs在不同门类中普遍存在,但其保守程度和系统发育信号(垂直遗传vs.水平转移)因CF和分类群而异,揭示了其进化历史的多样性。
10.定植模块在物种内呈现"全有或全无"的分布模式通过分析CMs在同一个物种不同菌株中的存在/缺失频率,发现CMs在物种内呈双峰分布——即在绝大多数菌株中要么都存在,要么都缺失,这种高度保守性解释了为何仅在近缘物种间比较难以发现它们。
11.YigZ的自然等位基因变异导致定植表型差异通过探究同一物种内(大肠杆菌)CFs的序列变异是否与定植能力差异相关,发现YigZ蛋白的关键氨基酸替换(如M25L,H146S)能显著影响定植效率,并且强定植等位基因在人体分离株中更常见,证明了微小的序列变异足以驱动生态适应。
相关新闻推荐
3、平湖红曲黄酒酿造过程中真菌菌群动态、红曲霉生长抑制因素(一)
